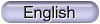2003年7月26日、重症急性呼吸器症候群(SARS)を引き起こすコロナウイルス(SARSウイルス)の、蛋白分解酵素(プロテアーゼ)の立体構造が、論文発表に先立ち蛋白立体構造の公的データベースであるProtein Data Bank(PDB)上に公開されました。
インシリコサイエンス社では、SARSの立体構造を予測、無償提供を行っております。X線解析の結果公開を受けまして、予測の精度を実証するために弊社でモデリングしたデータとの構造比較を行いました。(X線解析の座標に対してモデリングデータを合わせ、RMSを計算し精度を求めております。)
|
SARSモデリングデータとX線解析により求められた構造(PDB ID:1Q2W,July 26,2003)との比較(精度をRMSによって評価)(August 7,2003)
|
|
構造評価の画像
|

|
|

|
L1:Active site region
RMS= 1.03 A
|
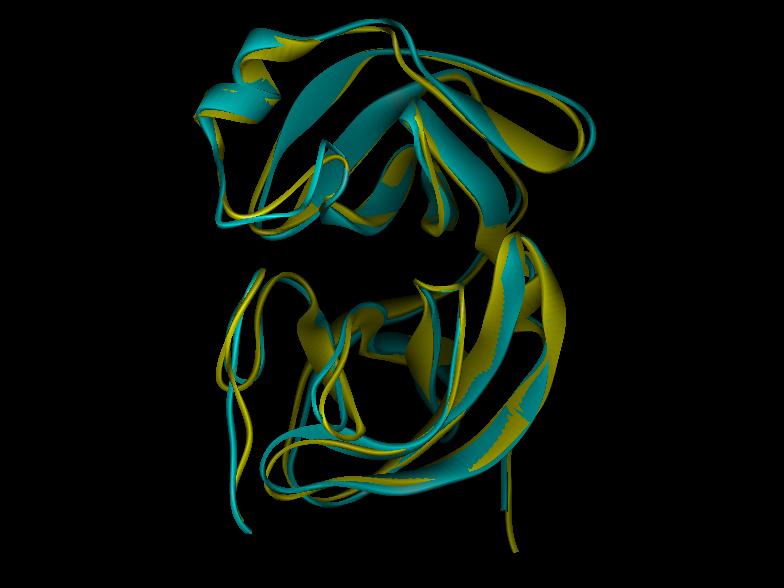
L2:Proteinase domain
RMS= 1.18 A
|
L3:Helix domain
RMS= 1.74 A
|
|
|
L4:SARS proteinase
RMS= 2.55 A
|
L5:SARS proteinase dimer
RMS= 2.82 A
|
|
ORF
|
1ab
|
|
protein_id
|
AAP13442.1
|
活性部位において、X線解析に対するずれはrmsで1.04Aと非常に精度良く予測が出来ていることがわかります。このことより弊社のモデリングデータはドラッグデザインに適切なモデルであったことが示されました。
|
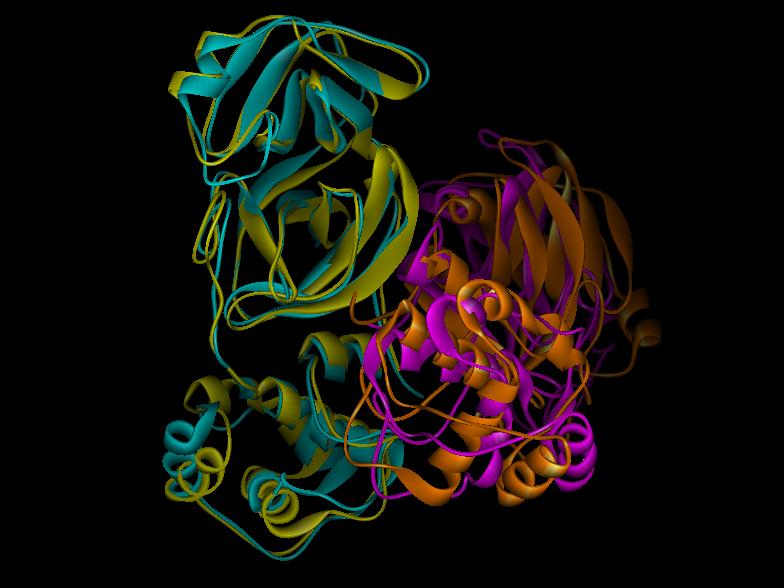
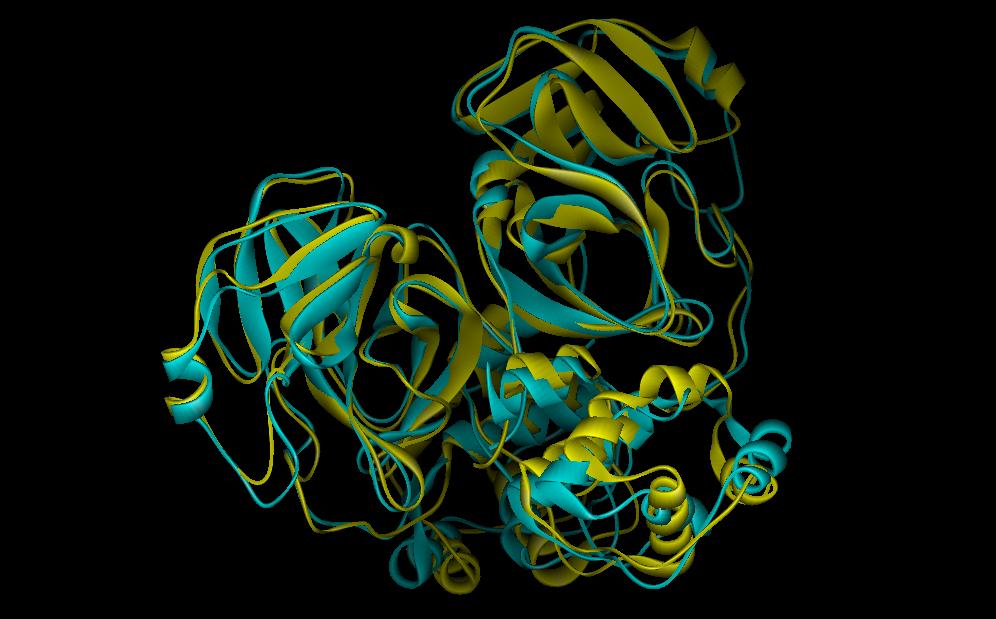
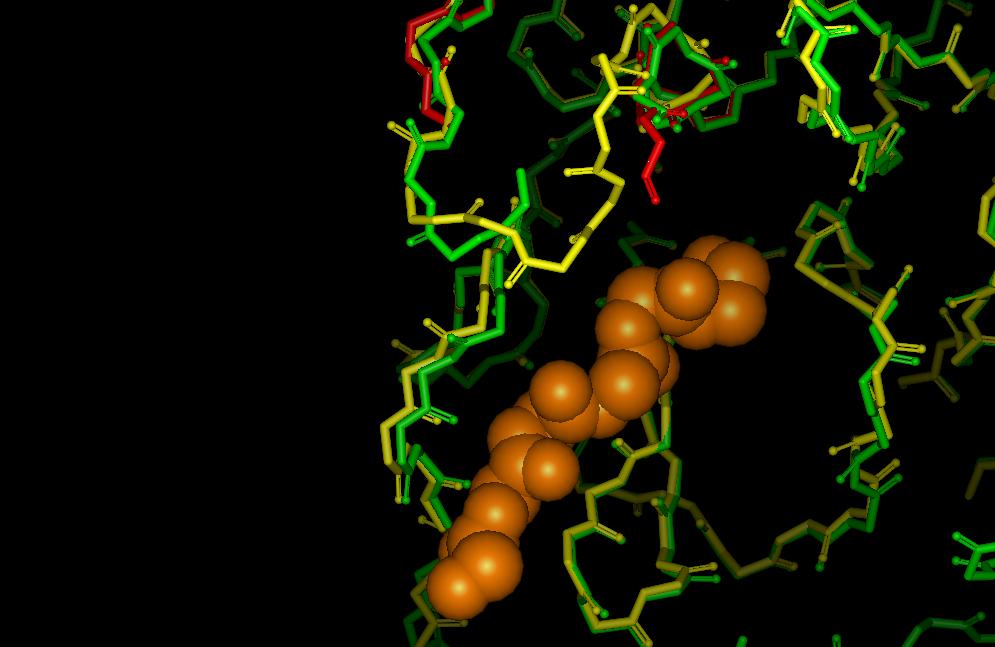
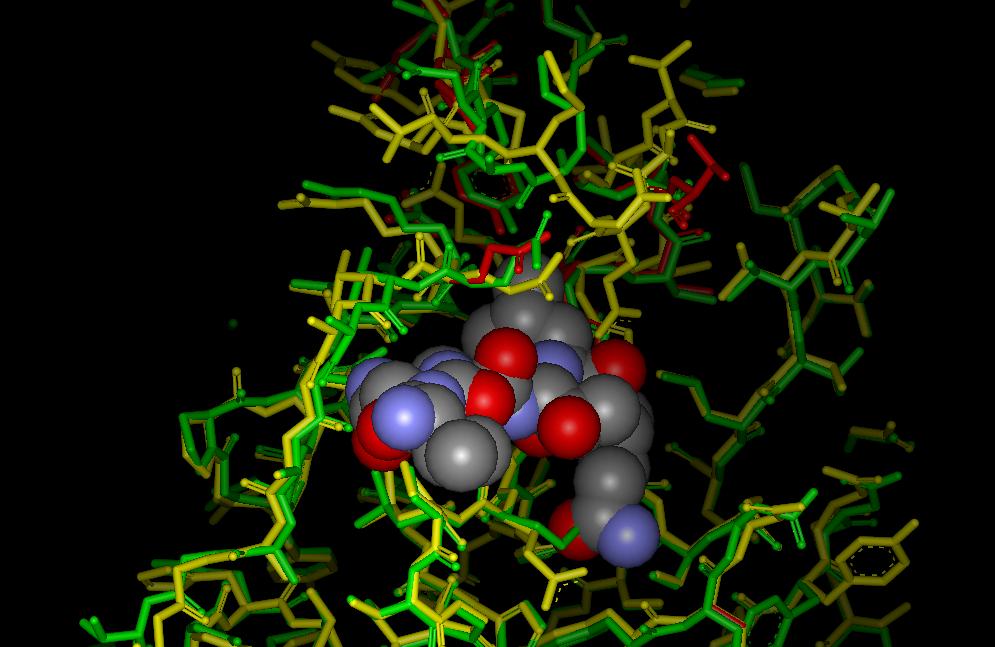
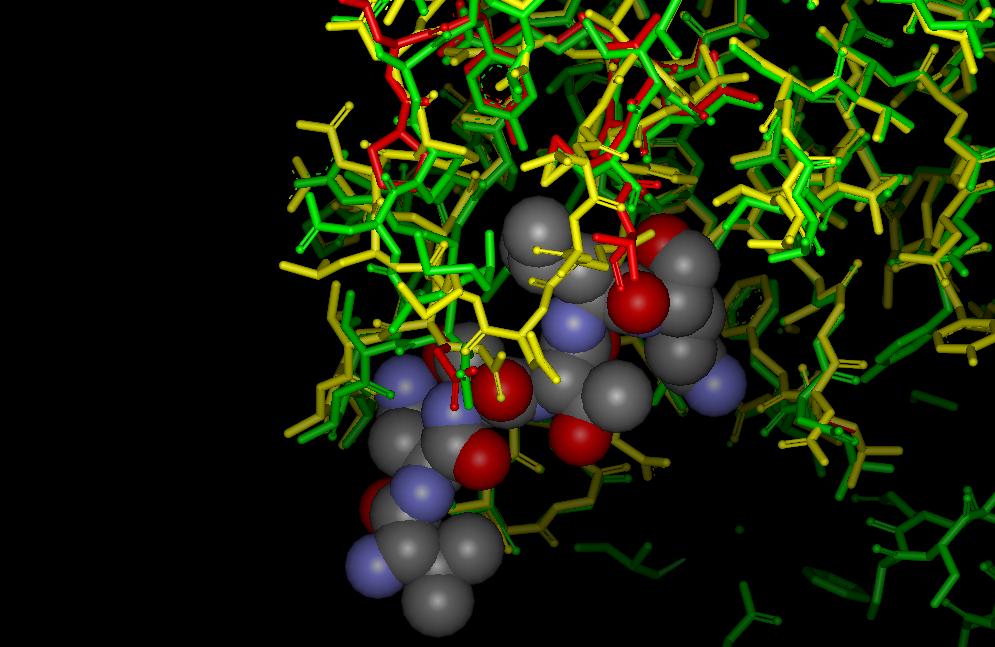
SARSプロテアーゼのX線解析により求められた構造(1Q2W :A鎖 緑, B鎖 赤)とFAMS Ligand&Complexで構築したモデル(黄) の比較およびリガンド(CPK)
|
|
構造評価の画像
|

|

|
|
|
主鎖のみ
|
|

|
|
|
側鎖付き
|
|
ORF
|
1ab
|
|
protein_id
|
AAP13442.1
|
さらに、比較を行いますとPDBに登録されたX線解析データ(PDB ID:1Q2W)には座標欠損があり、リガンド結合部位が正確に再現されていないことがわかります。それに対しPDFAMSシリーズで構築したモデルに関しましては結合部位も再現されていることがわかります。(以下に詳細説明を記載いたします。)
(1)SARS proteinaseと約45%のホモロジーのあるコロナウイルスのX線解析から得られた座標には、ペプチド性リガンドが結合しており、そのリガンドを、今回発表されたSARS proteinaseのX線結果から得られたタンパク質構造に結合させた時、リガンドの一部は実験データで報告されていない空間の近傍にあることがわかります。
(2)SARS proteinaseのX線結果で報告されていない数残基をPDFAMSシリーズを用いたモデリングで補完しますと、リガンドが非常にうまく入る様子が表現できます。本来、薬物の入る鍵穴を利用しての、コンピュータによるハイスループットシミュレーションでは鍵穴によく当てはまる薬物を、エネルギーの安定な順番にリストアップします。
しかし、今回のSARS proteinaseX線解析で報告された鍵穴に相当する部分は、鍵穴に相当する大切な一部分が(1)で述べたように欠落しています。即ち、鍵穴が正確に決められていない、欠陥鍵穴になっています。
(3)もし、上記のような欠陥のある鍵穴に対して、薬物のコンピュータによるハイスループットシミュレーションを行うと、本来なら、見つかるべき薬物の大きさより大きな(酵素の欠陥鍵穴座標部分を占有するような)、薬物がスクリーニングされてしまいます。
(4)従って、今回発表されたSARS proteinaseのX線結果のタンパク質座標をそのまま使用すると、間違ったスクリーニングをすることになってしまいます。
(5)薬物設計を行うにあたっては、SARS proteinaseのX線結果のタンパク質座標の欠損部分を、ホモロジーモデリングで補い、補完して完全なタンパク質座標をつくらねばなりません。要するに、SARS proteinaseのX線結果のタンパク質座標が欠損している部分に対して、ホモロジーモデリングを利用して補完する必要があるということです。上記に示した図はSARS proteinaseの補完モデルとなっております。
(6)この結果から今回発表されたSARS proteinaseのX線解析データは、そのままでは薬物設計に適さないということになります。つまり、さらに完全な状態のX線解析が必要であると考えられます。
X線解析データとの比較により、インシリコサイエンス社で作成したSARSモデリングデータは信頼できることが実証され、ドラッグデザインに弊社モデルを利用することも有用である点も実証されました。